近日,物理学院生物物理研究所赵蕴杰教授课题组在软凝聚态与生物物理研究中取得重要进展。相关成果以“RNA-Protein Interaction Prediction Using Network-Guided Deep Learning”为题,于2025年2月16日发表在自然系列期刊Communications Biology(中科院一区Top)上。赵蕴杰教授为该论文的通讯作者,2023级博士研究生刘浩泉为该论文的第一作者,华中师范大学为该论文的唯一通讯作者单位。
RNA复杂系统的相关理论研究是理论物理基础问题及其前沿交叉领域研究的重要方向之一。例如,RNA-蛋白质复合物在基因转录和转录后调控等关键细胞过程中发挥着重要作用,其功能的实现依赖于RNA与蛋白质之间复杂的物理相互作用,包括范德华力、静电相互作用、氢键等多种作用机制。然而,RNA与蛋白质间相互作用存在着诸多复杂特征,如RNA分子结构的高度灵活性、动态多变的结合模式,再加上高质量实验数据的匮乏,长久以来严重制约了对其进行精准预测的能力。发展精确高效的RNA-蛋白质相互作用预测理论计算新方法是当前研究的重要挑战和迫切需求。
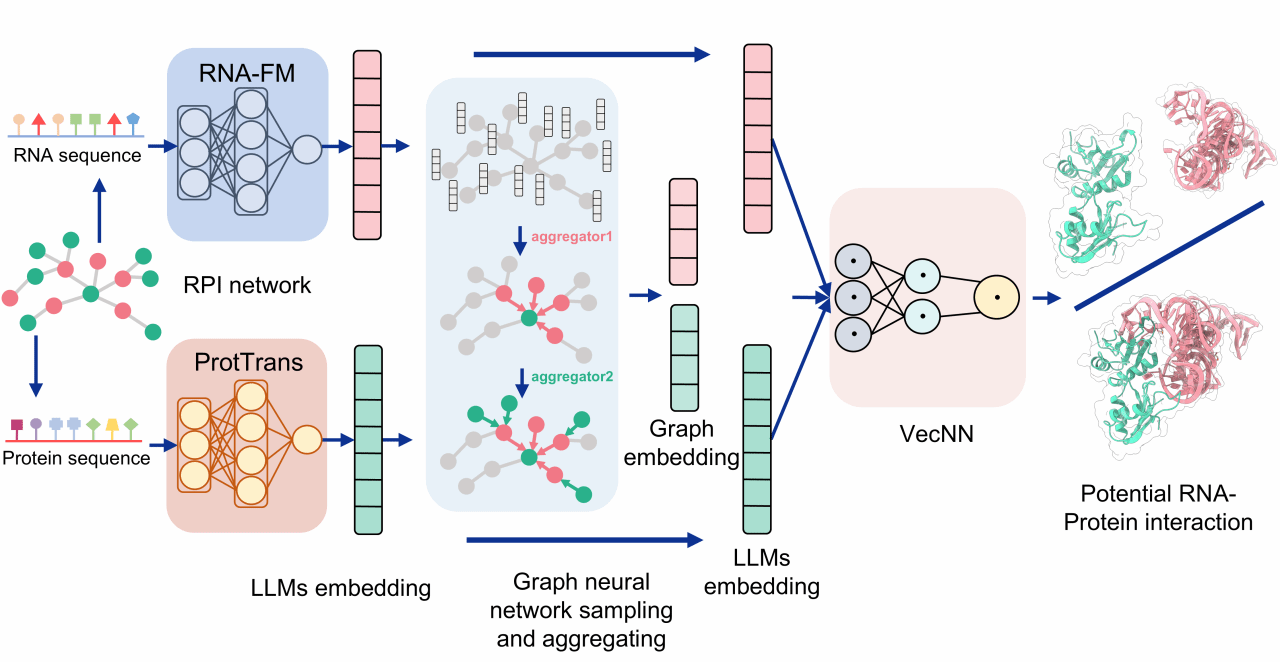
图1. ZHMolGraph模型流程图
针对这一科学难题,研究团队提出了ZHMolGraph方法。该方法将统计物理中的复杂网络模型与人工智能有机融合,为RNA-蛋白质相互作用(RPI)预测提供了新思路 (图1)。研究团队首先从大分子整体和残基两个层面出发对RPI网络进行了系统表征,揭示了RPI网络分布的不平衡性和模块化特征。在此基础上,ZHMolGraph模型融合了图神经网络和无监督大语言模型(LLMs),有效解决了现有RPI网络中的注释不平衡问题。一方面图神经网络能够有效捕捉RNA-蛋白质相互作用网络中无标度特性和复杂拓扑结构,而 LLMs 则擅长挖掘隐藏在序列信息背后的进化规律和功能模式,显著提升了对未知RNA和蛋白质的泛化能力。
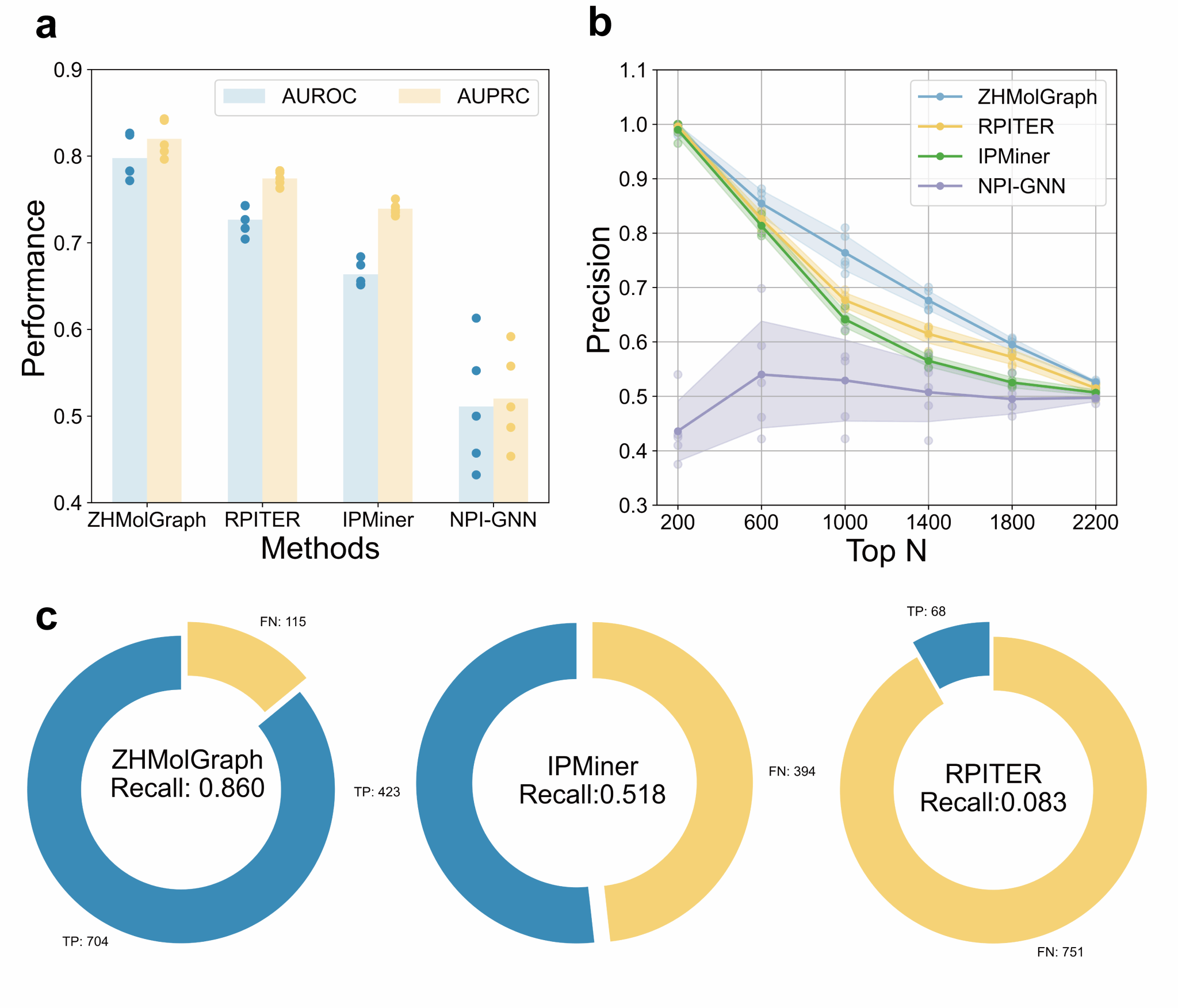
图2.未知RNA-蛋白质相互作用预测性能评估。
a, b 在未知RNA和蛋白质数据集TheNovel上,ZHMolGraph的平均AUROC和AUPRC(图a)和预测概率排序精确度(图b)显著优于现有方法。c SARS-CoV-2相关RNA-蛋白质相互作用预测结果表明,ZHMolGraph的召回率显著优于现有方法。
预测未知RNA和蛋白质间的相互作用物理规律是RNA-蛋白质相互作用预测中重要的科学问题。ZHMolGraph在模型未知的分子测试集上进行了评估,AUROC和AUPRC分别达到79.8%和82.0%。与现有方法相比,ZHMolGraph在AUROC指标上提升了7.1%-28.7%,在AUPRC指标上提升了4.6%-30.0% (图2a, b)。特别是在SARS-CoV-2相关病毒RNA-蛋白质相互作用的预测中,ZHMolGraph的召回率比现有最佳方法提高了34.2%,展现出对病毒物理机理识别的潜力(图2c)。这种一致的改进使ZHMolGraph成为一种可靠的选择,能够在不依赖实验结构的情况下确定RNA-蛋白质相互作用。ZHMolGraph是软凝聚态与生物物理研究领域中研究生物大分子的有效工具,将对RNA相关复合物结构设计与药物开发有重要意义。
课题组聚焦于RNA复杂系统的物理理论模型与智能计算方法的创新,在统计物理与人工智能融合领域取得了系列进展,相关前期研究成果已发表于Nature Communications,Briefings in Bioinformatics和Biophysics Reviews等生物物理领域的高水平学术期刊。本研究工作得到了国家自然科学基金面上项目、湖北省杰出青年基金和中央高校基本科研业务费等项目的资助。研究成果获得了中国生物物理学会“科技前沿”和SPRINGER NATURE旗下“Behind the Paper”等栏目的报道。
论文详见:https://www.nature.com/articles/s42003-025-07694-9
华大物院党委融媒体中心
华大物院团委宣传中心
通讯员|刘浩泉
责 编|孟晨轩
审 校|赵蕴杰 王 东
